株式会社ディー・エヌ・エー(所在地:東京都渋谷区、代表取締役社長兼CEO:岡村 信悟、以下DeNA)の子会社である株式会社DeNAライフサイエンス(本社:東京都渋谷区、代表取締役社長:砂田 真吾、以下 DeNAライフサイエンス)と東北大学東北メディカル・メガバンク機構(所在地:宮城県仙台市、機構長:山本 雅之、以下ToMMo)は、ToMMoが解析した日本人集団 の全ゲノムデータ(参照パネル)、および人工知能(AI)技術を活用した全ゲノムの遺伝型を推定する技術「遺伝子型インピュテーション」(以下 インピュテーション)の、別ゲノムコホート※1における有用性について共同で研究を行い、その成果を第68回日本人類遺伝学会にて発表しました。
研究の背景
限られた遺伝情報を安価に取得できるSNPアレイ※2解析のデータから、日本人集団がゲノム全域で保持する数千万におよぶ遺伝子多型の遺伝型を推定するインピュテーションは、高解像度な遺伝要因の探索や高精度な遺伝率および疾患発症リスクの推定を可能とするゲノム研究において有用な解析技術です。
日本人集団を対象とした高精度なインピュテーションには、日本人集団からなる多人数の全ゲノムデータ(参照パネル)が必要です。ToMMoでは、2018年に3,500人以上の日本人集団の参照パネル(以下3.5KJPNv2)※3を構築し、日本国内の地域住民を対象としたゲノムコホートにおいて高い精度を確認しています。しかし、異なるSNPアレイを用いる別ゲノムコホートにおける3.5KJPNv2の汎用性は十分に検証されていませんでした。
また、ToMMoでは参照パネルを用いることなく、個人情報保護に配慮してAI技術を応用したインピュテーション手法「RNN-IMP(Recurrent Neural Network – IMPutation)法※4」を開発し、シミュレーションにより高い精度を確認しました。このRNN-IMP法について、実際のSNPアレイデータを用いたさらなる検証が必要とされていました。
研究のねらい
今回、10万人以上のゲノムコホートであるMYCODEのSNPアレイ※5データに対して、3.5KJPNv2を用いたインピュテーションを行いその精度解析により、ToMMoとは別のゲノムコホートでの3.5KJPNv2の汎用性を検証します。
また、実際のSNPアレイデータに基づくRNN-IMP法の精度についても検証します。
研究結果
本研究により、3.5KJPNv2を用いることで比較的安価なSNPアレイデータから高精度なゲノム解析が実現できることを別ゲノムコホートで確認しました。また、RNN-IMP法の高い精度を実際のSNPアレイデータを用いて初めて確認し、個人情報の保護と利便性を両立した高精度のゲノム解析の実現性を示し、この研究成果を第68回日本人類遺伝学会にて発表しました。
1.3.5KJPNv2のMYCODEのSNPアレイデータに対する精度の高さを確認し、安価かつ高精度のゲノム解析の実現性を示す
MYCODE Researchの元で本研究の参加に同意した1,596名を対象に、インピュテーションの精度を検証しました。
その結果、3.5KJPNv2は、現在広く用いられている多人種集団の公開参照パネル(1KGP)※6と比較して、推定により復元されるSNP数が2.5倍程度増加し、またインピュテーション精度が最大190%向上※7しました(図)。
これにより、ToMMoとは別のSNPアレイを用いるMYCODEのゲノムコホートにおいても、3.5KJPNv2を用いたインピュテーションにより、安価で高精度なゲノム研究が実現できることが示されました。
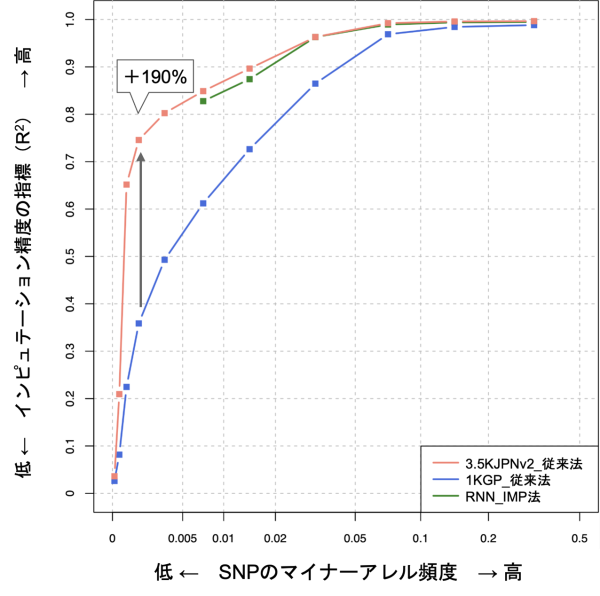
図 インピュテーション精度の比較結果
また、ToMMoで開発された日本人集団に最適化されたSNPアレイ「ジャポニカアレイ※8」とMYCODEのSNPアレイのインピュテーション精度への影響を比較したところ、日本人集団に一般的にみられるSNPについては同程度の精度を確認しました。
この結果は、今後MYCODEのデータとToMMoのデータを統合した大規模なゲノム研究の実施が技術的に可能であることを示しています。
2.RNN-IMP法の高い精度を実際のMYCODEのSNPアレイデータを用いて初めて確認し、個人情報の保護と利便性を両立した高精度のゲノム解析の実現性を示す
RNN-IMP法により3.5KJPNv2の全ゲノムデータをあらかじめ学習し、MYCODEのSNPアレイデータに対してインピュテーションを行いました。その結果、従来法と遜色なく高い精度が実現できることを、実際のSNPアレイデータを用いて初めて示しました(図)。この結果は、3.5KJPNv2にアクセスできない解析環境でも、個人情報の集合体である参照パネルを使わずにインピュテーションの精度向上が実現できることを示しています。
今後もDeNAライフサイエンスとToMMoは、日本におけるゲノム研究の発展を目指し、研究を続けていきます。
用語説明等
【MYCODE Researchについて】
DeNAライフサイエンスが行っているゲノム研究プロジェクト「MYCODE Research」では、一般向け遺伝子検査サービス「MYCODE」の会員約12万人のうち、約9割の方に研究参加同意をいただいており、インターネットを活用することでユーザーコミュニティの個人が自らの同意の下で研究に参加して科学の発展に寄与できる“Community-derived science”を実現しています。開始以来これまで累計約40件のアカデミアや企業との共同研究を実施しています。
※1 ゲノムコホート:ある特定の人々の集団を一定期間にわたって前向きに追跡し、遺伝型と疾病発症との関係を解析する研究などを行う対象集団。
※2 SNPアレイ:全ゲノム配列上に存在する一塩基多型(Single Nucleotide Polymorphism:SNP)を主としたDNA配列の個人差を計測する手法。全ゲノム配列を全て解析するより安価に計測が可能。
※3 日本人集団の参照パネル(3.5KJPNv2):ToMMoは岩手医科大学いわて東北メディカル・メガバンク機構と協力し地域住民 15 万人のコホート調査を 2013 年から実施している。この調査の参加者の全ゲノムを解析した結果に基づく日本人集団の参照パネル(全ゲノムリファレンスパネル)を規模を拡大しながら順次発表している。2018年6月に本研究でインピュテーションに用いた3.5KJPNv2(約3.5千人分)を、直近では2023年6月に54KJPN(約5.4万人分)を発表した。
参考:6.9万人分の全ゲノム情報の解析を完了し54KJPNを公開しました(ToMMoウェブサイト)
※4 RNN-IMP法:AI技術の一つである深層学習技術を利用することで従来の手法で用いられていた参照パネルの代わりに個人識別が困難な数値パラメータ情報を用いる形で、遺伝子型インピュテーションをする手法。参考論文;DOI:10.1371/journal.pcbi.1008207
※5 MYCODEのSNPアレイ;illumina社のSNPアレイをベースとして一部をカスタム設計し、70万箇所以上のSNPを搭載したSNPアレイ。
※6 公開参照パネル(1KGP):複数の民族集団から構成される 1,000 を越える検体を対象とした、全ゲノム配列情報の計測と遺伝子変異情報の網羅的な解析を行う国際研究プロジェクト「1000人ゲノムプロジェクト(1KGP)」より公開されている、2,504 検体についての全ゲノム配列データ。
※7 インピュテーション精度が最大190%向上:インピュテーション精度の指標として、一般的に、インピュテーションで推定された遺伝型と真の遺伝型の相関係数を2乗した値(R2)が用いられます。インピュテーションは推定するSNPのマイナーアレル頻度(例えば、あるSNPがAかCのいずれかのアレルをとる場合、集団の中で観測数の低い方のアレルの頻度)によって変化し、頻度の低いSNPは一般にインピュテーションが難しく、R2が低下する傾向があります。今回の結果では、参照パネルとして1KGPを用いた場合に頻度の低い領域で低下するR2が、3.5KJPNv2を用いた場合に190%の向上が確認されました。
※8 ジャポニカアレイ:66万箇所のSNPを搭載した日本人に最適化された初のSNPアレイ。
The post 全ゲノム遺伝型推定技術の有用性をMYCODE Researchで確認 ~DeNAライフサイエンスと東北メディカル・メガバンク機構が第68回日本人類遺伝学会で発表~ first appeared on 東北メディカル・メガバンク機構.